AlwaysR, Módulo III: Estadística en R
Clase 4: Modelos lineales II. Efectos aleatorios.
Dr. Giancarlo M. Correa

Modelos lineales II
Problemas con datos
Multicolinealidad
No genera problemas estadísticos, sin embargo, genera problemas en la interpretación.
No tiene sentido interpretar el efecto de X1 manteniendo otras variables independientes constante porque nosotros hemos observado una relación entre X1 y otras variables.
Problemas con datos
Multicolinealidad
No genera problemas estadísticos, sin embargo, genera problemas en la interpretación.
No tiene sentido interpretar el efecto de X1 manteniendo otras variables independientes constante porque nosotros hemos observado una relación entre X1 y otras variables.
head(seatpos)## Age Weight HtShoes Ht Seated Arm Thigh Leg hipcenter## 1 46 180 187.2 184.9 95.2 36.1 45.3 41.3 -206.300## 2 31 175 167.5 165.5 83.8 32.9 36.5 35.9 -178.210## 3 23 100 153.6 152.2 82.9 26.0 36.6 31.0 -71.673## 4 19 185 190.3 187.4 97.3 37.4 44.1 41.0 -257.720## 5 23 159 178.0 174.1 93.9 29.5 40.1 36.9 -173.230## 6 47 170 178.7 177.0 92.4 36.0 43.2 37.4 -185.150mod2 = lm(hipcenter ~ ., data = seatpos)summary(mod2)## ## Call:## lm(formula = hipcenter ~ ., data = seatpos)## ## Residuals:## Min 1Q Median 3Q Max ## -73.827 -22.833 -3.678 25.017 62.337 ## ## Coefficients:## Estimate Std. Error t value Pr(>|t|) ## (Intercept) 436.43213 166.57162 2.620 0.0138 *## Age 0.77572 0.57033 1.360 0.1843 ## Weight 0.02631 0.33097 0.080 0.9372 ## HtShoes -2.69241 9.75304 -0.276 0.7845 ## Ht 0.60134 10.12987 0.059 0.9531 ## Seated 0.53375 3.76189 0.142 0.8882 ## Arm -1.32807 3.90020 -0.341 0.7359 ## Thigh -1.14312 2.66002 -0.430 0.6706 ## Leg -6.43905 4.71386 -1.366 0.1824 ## ---## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1## ## Residual standard error: 37.72 on 29 degrees of freedom## Multiple R-squared: 0.6866, Adjusted R-squared: 0.6001 ## F-statistic: 7.94 on 8 and 29 DF, p-value: 1.306e-05Problemas con datos
La matriz de correlación entre todas las variables:
round(cor(seatpos), digits = 2)## Age Weight HtShoes Ht Seated Arm Thigh Leg hipcenter## Age 1.00 0.08 -0.08 -0.09 -0.17 0.36 0.09 -0.04 0.21## Weight 0.08 1.00 0.83 0.83 0.78 0.70 0.57 0.78 -0.64## HtShoes -0.08 0.83 1.00 1.00 0.93 0.75 0.72 0.91 -0.80## Ht -0.09 0.83 1.00 1.00 0.93 0.75 0.73 0.91 -0.80## Seated -0.17 0.78 0.93 0.93 1.00 0.63 0.61 0.81 -0.73## Arm 0.36 0.70 0.75 0.75 0.63 1.00 0.67 0.75 -0.59## Thigh 0.09 0.57 0.72 0.73 0.61 0.67 1.00 0.65 -0.59## Leg -0.04 0.78 0.91 0.91 0.81 0.75 0.65 1.00 -0.79## hipcenter 0.21 -0.64 -0.80 -0.80 -0.73 -0.59 -0.59 -0.79 1.00Problemas con datos
¿Qué podemos hacer cuando tenemos variables correlacionadas?
Seleccionar las variables más importantes
Identificar las variables correlacionadas claves para el modelo: variance inflation factors (VIF)
round(vif(mod2), digits = 2)## Age Weight HtShoes Ht Seated Arm Thigh Leg ## 2.00 3.65 307.43 333.14 8.95 4.50 2.76 6.69Valores VIFj>10 indican una alta colinealidad.
Problemas con datos
¿Qué podemos hacer cuando tenemos variables correlacionadas?
Seleccionar las variables más importantes
Identificar las variables correlacionadas claves para el modelo: variance inflation factors (VIF)
round(vif(mod2), digits = 2)## Age Weight HtShoes Ht Seated Arm Thigh Leg ## 2.00 3.65 307.43 333.14 8.95 4.50 2.76 6.69Valores VIFj>10 indican una alta colinealidad.
Eliminar algunas variables no significa que ellas no están asociadas con la variable respuesta, solo que no las necesitamos para predecir la respuesta.
Datos faltantes
Podemos hablar de dos casos:
Caso faltante: cuando no tenemos información de todas las variables en una observación en el conjunto de datos.
Valores faltantes: cuando no tenemos información de algunas variables en una observación en el conjunto de datos.
head(chmiss, n = 10)## race fire theft age involact income## 60626 10.0 6.2 29 60.4 NA 11.744## 60640 22.2 9.5 44 76.5 0.1 9.323## 60613 19.6 10.5 36 NA 1.2 9.948## 60657 17.3 7.7 37 NA 0.5 10.656## 60614 24.5 8.6 53 81.4 0.7 9.730## 60610 54.0 34.1 68 52.6 0.3 8.231## 60611 4.9 11.0 75 42.6 0.0 21.480## 60625 7.1 6.9 18 78.5 0.0 11.104## 60618 5.3 7.3 31 90.1 NA 10.694## 60647 21.5 15.1 NA 89.8 1.1 9.631Datos faltantes
Hay dos enfoques para lidiar con esto:
Métodos de supresión: eliminar las observaciones con algun tipo de datos faltantes.
Métodos de retención: retienen las observaciones con datos faltantes para implementar una regresión modificada para datos faltantes.
Método de supresión
Simplemente eliminar las observaciones con datos faltantes e implementar el modelo luego. R hace esto por nosotros automaticamente.
## race fire theft age involact income## 60626 10.0 6.2 29 60.4 NA 11.744## 60640 22.2 9.5 44 76.5 0.1 9.323## 60613 19.6 10.5 36 NA 1.2 9.948## 60657 17.3 7.7 37 NA 0.5 10.656## 60614 24.5 8.6 53 81.4 0.7 9.730## 60610 54.0 34.1 68 52.6 0.3 8.231## 60611 4.9 11.0 75 42.6 0.0 21.480Método de retención
Imputación: completar los datos perdidos con la media de cada variable. Se puede usar también la mediana, moda, etc. Problemas: Introduce error. Métodos de estimación de parámetros pueden estar sesgados.
Imputación a partir de regresión: Implementar un modelo lineal con las observaciones completas. Luego, predecir valores cuando hayan datos faltantes. En este caso, la variable respuesta será la variable que tiene datos faltantes y las variables independientes todas las demás variables.
Selección de modelos
El proceso de seleccionar tan pocas variables como sea posible para incluir en el modelo lineal (principio de parsimonia).
head(case1201)## State SAT Takers Income Years Public Expend Rank## 1 Iowa 1088 3 326 16.79 87.8 25.60 89.7## 2 SouthDakota 1075 2 264 16.07 86.2 19.95 90.6## 3 NorthDakota 1068 3 317 16.57 88.3 20.62 89.8## 4 Kansas 1045 5 338 16.30 83.9 27.14 86.3## 5 Nebraska 1045 5 293 17.25 83.6 21.05 88.5## 6 Montana 1033 8 263 15.91 93.7 29.48 86.4mod3 = lm(SAT ~ Takers + Income + Years + Public + Expend + Rank, data=case1201)summary(mod3)## ## Call:## lm(formula = SAT ~ Takers + Income + Years + Public + Expend + ## Rank, data = case1201)## ## Residuals:## Min 1Q Median 3Q Max ## -60.046 -6.768 0.972 13.947 46.332 ## ## Coefficients:## Estimate Std. Error t value Pr(>|t|) ## (Intercept) -94.659109 211.509584 -0.448 0.656731 ## Takers -0.480080 0.693711 -0.692 0.492628 ## Income -0.008195 0.152358 -0.054 0.957353 ## Years 22.610082 6.314577 3.581 0.000866 ***## Public -0.464152 0.579104 -0.802 0.427249 ## Expend 2.212005 0.845972 2.615 0.012263 * ## Rank 8.476217 2.107807 4.021 0.000230 ***## ---## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1## ## Residual standard error: 26.34 on 43 degrees of freedom## Multiple R-squared: 0.8787, Adjusted R-squared: 0.8618 ## F-statistic: 51.91 on 6 and 43 DF, p-value: < 2.2e-16Selección de modelos
Con esta base de datos, el modelo más simple que podemos implementar es:
SATi=β0+ϵi
El modelo más complicado es:
SATi=β0+β1Incomei+β2Takersi+β3Yearsi+β4Ranki+β5Expendi+β6Publici+ϵi
¿Cuantos modelos podemos implementar con esta base de datos?
26 modelos posibles! (sin considerar interacciones!)
Selección de modelos
Eliminación hacia atrás: Comienza con el full model. Elimina la variable con mayor p-value y corre el modelo nuevamente. Repite este proceso hasta que el modelo solo contiene variables con p-value por dejabo de nivel de significancia α.
Selección hacia adelante: Comienza con el modelo más sencillo (solo contiene intercepto). Añade la variable con menor p-value por debajo de nivel de significancia α. Repetir hasta que no haya variable que pueda ser añadida con p-value por debajo de nivel de significancia α.
Selección de modelos
Eliminación hacia atrás: Comienza con el full model. Elimina la variable con mayor p-value y corre el modelo nuevamente. Repite este proceso hasta que el modelo solo contiene variables con p-value por dejabo de nivel de significancia α.
Selección hacia adelante: Comienza con el modelo más sencillo (solo contiene intercepto). Añade la variable con menor p-value por debajo de nivel de significancia α. Repetir hasta que no haya variable que pueda ser añadida con p-value por debajo de nivel de significancia α.
Limitaciones
- Inclusión en el modelo no necesariamente significa que la variable es importante.
- Exclusión no necesariamente significa que la variable no sea importante.
- Se tiende a elegir los modelos más pequeños que los óptimos para predicción.
mod3 = lm(SAT ~ Takers + Income + Years + Public + Expend + Rank, data=case1201)backmod = ols_step_backward_p(mod3, prem=0.05, progress=FALSE) # Seleccion hacia atrassummary(backmod$model)## ## Call:## lm(formula = paste(response, "~", paste(preds, collapse = " + ")), ## data = l)## ## Residuals:## Min 1Q Median 3Q Max ## -64.802 -6.798 2.169 17.525 49.706 ## ## Coefficients:## Estimate Std. Error t value Pr(>|t|) ## (Intercept) -303.7243 97.8415 -3.104 0.00326 ** ## Years 26.0952 5.3894 4.842 1.49e-05 ***## Expend 1.8609 0.6351 2.930 0.00526 ** ## Rank 9.8258 0.5987 16.412 < 2e-16 ***## ---## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1## ## Residual standard error: 26.25 on 46 degrees of freedom## Multiple R-squared: 0.8711, Adjusted R-squared: 0.8627 ## F-statistic: 103.6 on 3 and 46 DF, p-value: < 2.2e-16mod3 = lm(SAT ~ Takers + Income + Years + Public + Expend + Rank, data=case1201)forwardmod = ols_step_forward_p(mod3, penter=0.05, progress=FALSE) # Seleccion hacia adelantesummary(forwardmod$model)## ## Call:## lm(formula = paste(response, "~", paste(preds, collapse = " + ")), ## data = l)## ## Residuals:## Min 1Q Median 3Q Max ## -64.802 -6.798 2.169 17.525 49.706 ## ## Coefficients:## Estimate Std. Error t value Pr(>|t|) ## (Intercept) -303.7243 97.8415 -3.104 0.00326 ** ## Rank 9.8258 0.5987 16.412 < 2e-16 ***## Years 26.0952 5.3894 4.842 1.49e-05 ***## Expend 1.8609 0.6351 2.930 0.00526 ** ## ---## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1## ## Residual standard error: 26.25 on 46 degrees of freedom## Multiple R-squared: 0.8711, Adjusted R-squared: 0.8627 ## F-statistic: 103.6 on 3 and 46 DF, p-value: < 2.2e-16Selección de modelos
Métodos basados en criterio:
AIC: Criterio de información de Akaike. Toma en cuenta el número de observaciones, parámetros, y suma de residuales. Normalmente el menor AIC nos indica un mejor modelo.
BIC: Criterio de información Bayesiana. Toma en cuenta el número de observaciones, parámetros, y suma de residuales. Normalmente el menor BIC nos indica un mejor modelo.
R2−adj: El mayor valor nos indica el mejor modelo.
Selección de modelos
Métodos basados en criterio:
AIC: Criterio de información de Akaike. Toma en cuenta el número de observaciones, parámetros, y suma de residuales. Normalmente el menor AIC nos indica un mejor modelo.
BIC: Criterio de información Bayesiana. Toma en cuenta el número de observaciones, parámetros, y suma de residuales. Normalmente el menor BIC nos indica un mejor modelo.
R2−adj: El mayor valor nos indica el mejor modelo.
Si deseas obtener el mejor modelo, se recomienda usar BIC.
Si deseas usar el modelo para hacer predicciones, se recomienda usar AIC.
Los modelos no necesitan ser anidados para ser comparados (pero si deben contener los mismos datos).
Problemas con supuestos
Linealidad de la asociación
- Consecuencia depende en el grado de no-linealidad.
- Buscar alternativas (e.g. modelos aditivos generalizados, splines, etc.)
Independencia
- Un modelo lineal no es robusto a la no independencia de observaciones.
- Alternativa, usar la función
glsen caso de dependencia temporal (siempre y cuando se conozca la estructura del error).
Problemas con supuestos
Linealidad de la asociación
- Consecuencia depende en el grado de no-linealidad.
- Buscar alternativas (e.g. modelos aditivos generalizados, splines, etc.)
Independencia
- Un modelo lineal no es robusto a la no independencia de observaciones.
- Alternativa, usar la función
glsen caso de dependencia temporal (siempre y cuando se conozca la estructura del error).
Normalidad
- Si se tiene un muestreo grande n, este supuesto no causa problemas importantes.
- Se recomienda usar ejercicios de simulación para saber los efectos de la no-normalidad de residuos sobre los parámetros estimados.
Transformaciones
Pueden ayudar en el ajuste del modelo y cumplir los supuestos de la regresión lineal (e.g. supuesto de varianza constante).
En vez de Yi=β0+β1Xi+ϵi, podemos escribir:
log(Yi)=β0+β1Xi+ϵi
Despues de ajustar el modelo, tenemos que volver a la escala original:
Yi=exp(β0+β1Xi)exp(ϵi)
Transformaciones
Pueden ayudar en el ajuste del modelo y cumplir los supuestos de la regresión lineal (e.g. supuesto de varianza constante).
En vez de Yi=β0+β1Xi+ϵi, podemos escribir:
log(Yi)=β0+β1Xi+ϵi
Despues de ajustar el modelo, tenemos que volver a la escala original:
Yi=exp(β0+β1Xi)exp(ϵi)
Obtener la variable respuesta en su escala original no es muy complicado. Sin embargo, los coeficientes estimados ya no tienen la misma interpretación. Por ejemplo:
- Un incremento en una unidad en X1 ahora multiplicaría la respuesta media (en su escala original) por e^β1.
Transformaciones
Pueden ayudar en el ajuste del modelo y cumplir los supuestos de la regresión lineal (e.g. supuesto de varianza constante).
En vez de Yi=β0+β1Xi+ϵi, podemos escribir:
log(Yi)=β0+β1Xi+ϵi
Despues de ajustar el modelo, tenemos que volver a la escala original:
Yi=exp(β0+β1Xi)exp(ϵi)
Obtener la variable respuesta en su escala original no es muy complicado. Sin embargo, los coeficientes estimados ya no tienen la misma interpretación. Por ejemplo:
- Un incremento en una unidad en X1 ahora multiplicaría la respuesta media (en su escala original) por e^β1.
Para mayor información sobre transformaciones, ver aquí.
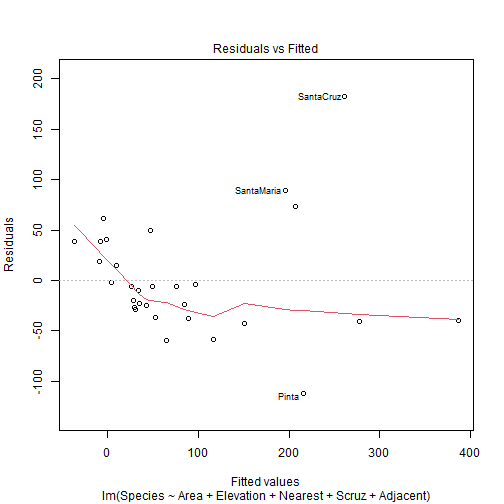
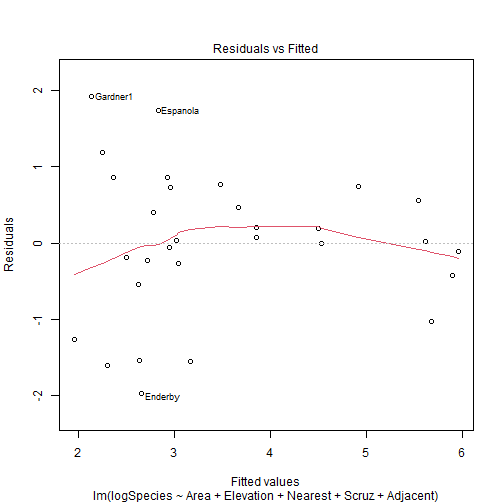
head(gala)## Species Endemics Area Elevation Nearest Scruz Adjacent## Baltra 58 23 25.09 346 0.6 0.6 1.84## Bartolome 31 21 1.24 109 0.6 26.3 572.33## Caldwell 3 3 0.21 114 2.8 58.7 0.78## Champion 25 9 0.10 46 1.9 47.4 0.18## Coamano 2 1 0.05 77 1.9 1.9 903.82## Daphne.Major 18 11 0.34 119 8.0 8.0 1.84Efectos aleatorios
Efectos aleatorios
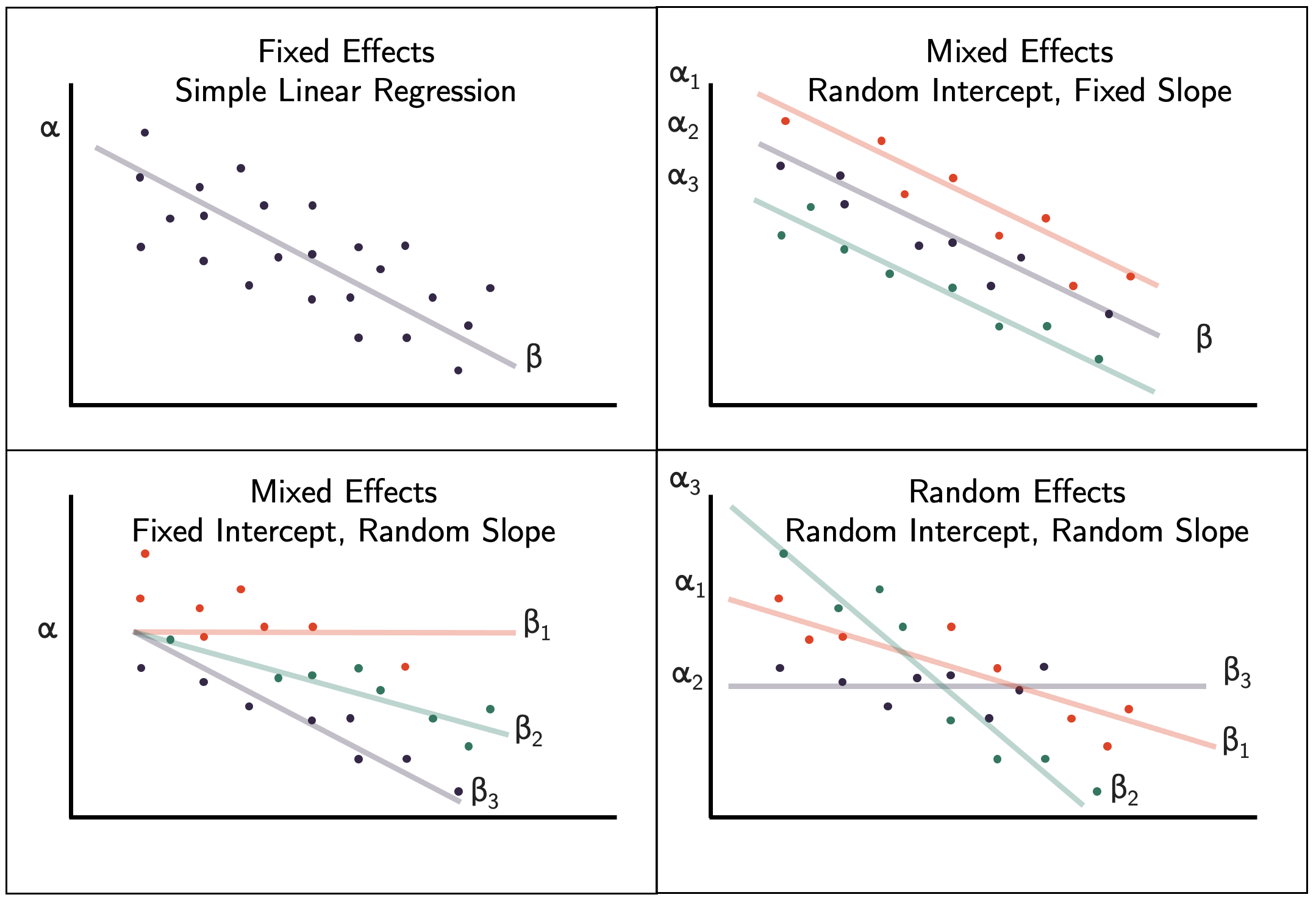
Modelo de efectos mixtos: cuando tenemos efectos fijos (como hemos visto hasta el momento) y efectos aleatorios (como describiremos aquí).
Efecto fijo: Son variables que esperamos tengan un efecto sobre la variable respuesta ( Y ). Es decir, es el X como lo hemos visto hasta el momento. Puede ser continua o categórica. Se asume que no hay relación entre los diferentes niveles de una variable. Ejemplo: un efecto para hembras y un efecto para machos, pero estos efectos no interactúan uno con el otro.
Efectos aleatorios
Modelo de efectos mixtos: cuando tenemos efectos fijos (como hemos visto hasta el momento) y efectos aleatorios (como describiremos aquí).
Efecto fijo: Son variables que esperamos tengan un efecto sobre la variable respuesta ( Y ). Es decir, es el X como lo hemos visto hasta el momento. Puede ser continua o categórica. Se asume que no hay relación entre los diferentes niveles de una variable. Ejemplo: un efecto para hembras y un efecto para machos, pero estos efectos no interactúan uno con el otro.
Efecto aleatorio: Son factores de agrupación que tratamos de controlar. Siempre son variables categóricas. Normalmente no estamos interesados en el efecto de esta variable sobre nuestra variable respuesta Y, pero sabemos que puede influir en nuestros resultados. Los niveles o grupos en un efecto aleatorio puede ser pensado como una muestra de niveles de una población más grande de niveles. Se recomienda un mínimo de 5 niveles en nuestros datos.
Efectos aleatorios
Tomar en cuenta:
- No usar efectos aleatorios cuando el número de niveles es bajo (3 o menor).
- No usar efectos aleatorios cuando no se quiere asumir que estos niveles vienen de una distribución común en la población.
¿En qué casos debo incorporar efectos aleatorios?
¿Los factores pueden ser considerados como una muestra aleatoria de una distribución de probabilidad?
¿La investigación tiene por objetivo hacer inferencia a una población mayor a la incluida en el modelo?
¿Hay una falta de independencia estadística debido a múltiples observaciones para un mismo nivel?
Efectos aleatorios
head(sleepstudy, n = 25)## Reaction Days Subject## 1 249.5600 0 308## 2 258.7047 1 308## 3 250.8006 2 308## 4 321.4398 3 308## 5 356.8519 4 308## 6 414.6901 5 308## 7 382.2038 6 308## 8 290.1486 7 308## 9 430.5853 8 308## 10 466.3535 9 308## 11 222.7339 0 309## 12 205.2658 1 309## 13 202.9778 2 309## 14 204.7070 3 309## 15 207.7161 4 309## 16 215.9618 5 309## 17 213.6303 6 309## 18 217.7272 7 309## 19 224.2957 8 309## 20 237.3142 9 309## 21 199.0539 0 310## 22 194.3322 1 310## 23 234.3200 2 310## 24 232.8416 3 310## 25 229.3074 4 310Un modelo con efecto aleatorio en el intercepto:
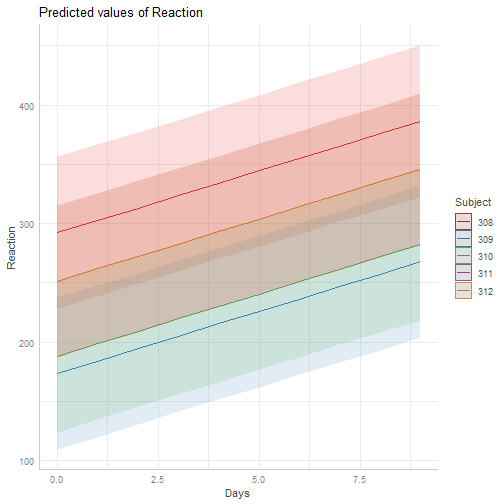
lmod1 = lmer ( Reaction ~ Days + ( 1 | Subject ) , data= sleepstudy )summary(lmod1)## Linear mixed model fit by REML ['lmerMod']## Formula: Reaction ~ Days + (1 | Subject)## Data: sleepstudy## ## REML criterion at convergence: 1786.5## ## Scaled residuals: ## Min 1Q Median 3Q Max ## -3.2257 -0.5529 0.0109 0.5188 4.2506 ## ## Random effects:## Groups Name Variance Std.Dev.## Subject (Intercept) 1378.2 37.12 ## Residual 960.5 30.99 ## Number of obs: 180, groups: Subject, 18## ## Fixed effects:## Estimate Std. Error t value## (Intercept) 251.4051 9.7467 25.79## Days 10.4673 0.8042 13.02## ## Correlation of Fixed Effects:## (Intr)## Days -0.371Un modelo con efecto aleatorio en el intercepto:
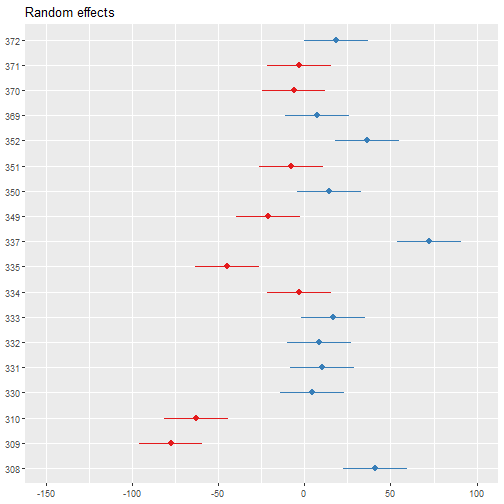
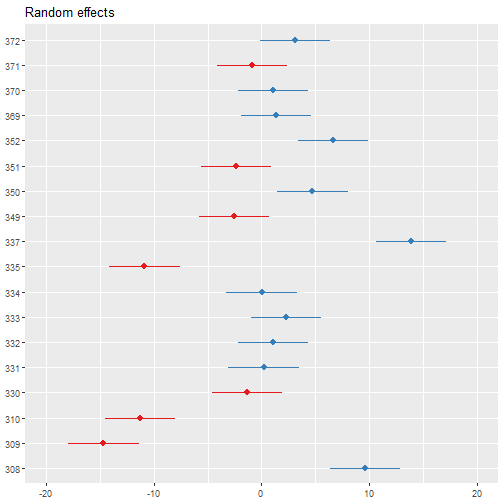
coef(lmod1)$Subject## (Intercept) Days## 308 292.1888 10.46729## 309 173.5556 10.46729## 310 188.2965 10.46729## 330 255.8115 10.46729## 331 261.6213 10.46729## 332 259.6263 10.46729## 333 267.9056 10.46729## 334 248.4081 10.46729## 335 206.1230 10.46729## 337 323.5878 10.46729## 349 230.2089 10.46729## 350 265.5165 10.46729## 351 243.5429 10.46729## 352 287.7835 10.46729## 369 258.4415 10.46729## 370 245.0424 10.46729## 371 248.1108 10.46729## 372 269.5209 10.46729Un modelo con efecto aleatorio en la pendiente:
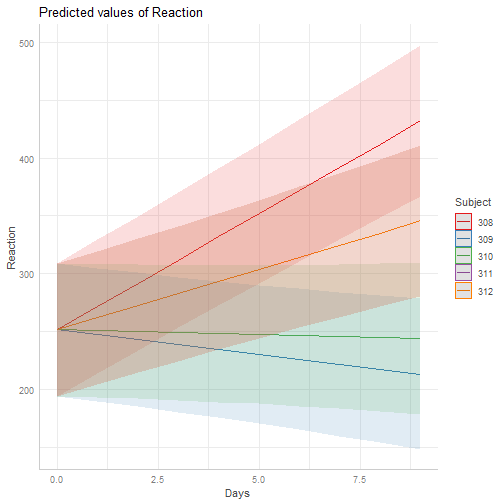
lmod2 = lmer ( Reaction ~ Days + ( 0 + Days | Subject ) , data= sleepstudy )summary(lmod2)## Linear mixed model fit by REML ['lmerMod']## Formula: Reaction ~ Days + (0 + Days | Subject)## Data: sleepstudy## ## REML criterion at convergence: 1766.5## ## Scaled residuals: ## Min 1Q Median 3Q Max ## -3.5104 -0.5588 0.0541 0.6244 4.6022 ## ## Random effects:## Groups Name Variance Std.Dev.## Subject Days 52.71 7.26 ## Residual 842.03 29.02 ## Number of obs: 180, groups: Subject, 18## ## Fixed effects:## Estimate Std. Error t value## (Intercept) 251.41 4.02 62.539## Days 10.47 1.87 5.599## ## Correlation of Fixed Effects:## (Intr)## Days -0.340Un modelo con efecto aleatorio en la pendiente:
coef(lmod2)$Subject## (Intercept) Days## 308 251.4051 20.0866918## 309 251.4051 -4.2326711## 310 251.4051 -0.8189202## 330 251.4051 9.1273878## 331 251.4051 10.6754843## 332 251.4051 11.5352979## 333 251.4051 12.7430088## 334 251.4051 10.4774867## 335 251.4051 -0.4337385## 337 251.4051 24.3577325## 349 251.4051 7.9069248## 350 251.4051 15.2012144## 351 251.4051 8.1041559## 352 251.4051 17.1349527## 369 251.4051 11.8340809## 370 251.4051 11.5298510## 371 251.4051 9.5898795## 372 251.4051 13.5923281Un modelo con efecto aleatorio en el intercepto y la pendiente:
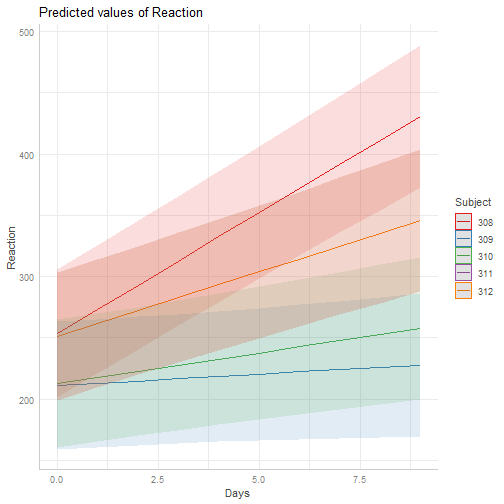
lmod3 = lmer ( Reaction ~ Days + ( Days | Subject ) , data= sleepstudy )summary(lmod3)## Linear mixed model fit by REML ['lmerMod']## Formula: Reaction ~ Days + (Days | Subject)## Data: sleepstudy## ## REML criterion at convergence: 1743.6## ## Scaled residuals: ## Min 1Q Median 3Q Max ## -3.9536 -0.4634 0.0231 0.4634 5.1793 ## ## Random effects:## Groups Name Variance Std.Dev. Corr## Subject (Intercept) 612.10 24.741 ## Days 35.07 5.922 0.07## Residual 654.94 25.592 ## Number of obs: 180, groups: Subject, 18## ## Fixed effects:## Estimate Std. Error t value## (Intercept) 251.405 6.825 36.838## Days 10.467 1.546 6.771## ## Correlation of Fixed Effects:## (Intr)## Days -0.138Un modelo con efecto aleatorio en el intercepto y la pendiente:
coef(lmod3)$Subject## (Intercept) Days## 308 253.6637 19.6662617## 309 211.0064 1.8476053## 310 212.4447 5.0184295## 330 275.0957 5.6529356## 331 273.6654 7.3973743## 332 260.4447 10.1951090## 333 268.2456 10.2436499## 334 244.1725 11.5418676## 335 251.0714 -0.2848792## 337 286.2956 19.0955511## 349 226.1949 11.6407181## 350 238.3351 17.0815038## 351 255.9830 7.4520239## 352 272.2688 14.0032871## 369 254.6806 11.3395008## 370 225.7921 15.2897709## 371 252.2122 9.4791297## 372 263.7197 11.7513080Resultados
sjPlot::tab_model(lmod1)| Reaction | |||
|---|---|---|---|
| Predictors | Estimates | CI | p |
| (Intercept) | 251.41 | 232.17 – 270.64 | <0.001 |
| Days | 10.47 | 8.88 – 12.05 | <0.001 |
| Random Effects | |||
| σ2 | 960.46 | ||
| τ00 Subject | 1378.18 | ||
| ICC | 0.59 | ||
| N Subject | 18 | ||
| Observations | 180 | ||
| Marginal R2 / Conditional R2 | 0.280 / 0.704 | ||
ICC cercano a cero nos sugiere que un modelo con efectos aleatorios no es necesario.